東京大学医科学研究所(以下、東大医科研) 附属ヒトゲノム解析センターと日本アイ・ビー・エム株式会社は、新型コロナウイルスの変異状況のモニタリングならびにウイルスの感染経路を同定するのに活用できる「HGC SARS-CoV-2 Variant Browser」をIBMと開発。スーパーコンピュータSHIROKANE上で運用を開始したことを発表しました。
「HGC SARS-CoV-2 Variant Browser」の開発で可能になること
現在、私たちの生活に大きな影響を与えている新型コロナウイルス。ゲノム変異を繰り返し、感染性が高まったり、ワクチンの効果を回避したりするような変異株の出現が懸念されるなど、新たな脅威が注目されています。
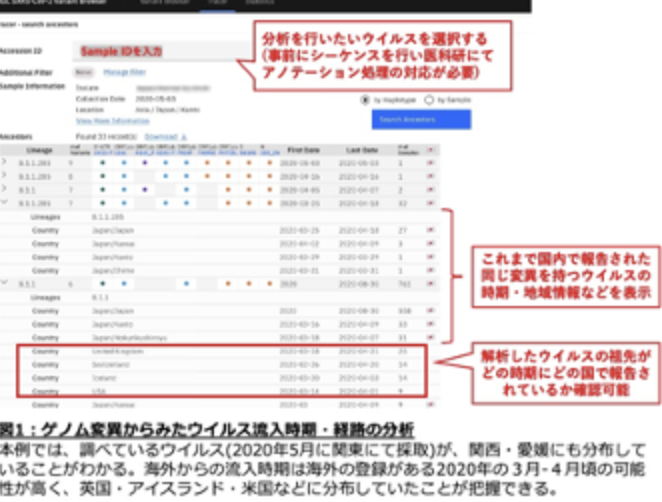
そのリスク軽減につながるとされているのが、ウイルスゲノムの解析。2021年5月時点で、世界中から集められた170万人以上の感染者から取得された新型コロナウイルスのゲノム配列が公開。これらの情報を活用すれば、感染経路の確定などにつながると考えられていたものの、これまでは膨大な数のウイルスゲノムを迅速に解析、結果を提供する体制は十分に整備できているとは言えない状況でした。
今回の開発は、このような研究体制の確立に大きな意味を与えるもの。HGC SARS-CoV-2 Variant Browserを活用することで、「海外からの流入時期ならびに感染経路推定」や「新型コロナウイルスの変異状況の把握」、「新型コロナウイルスのゲノムの国内登録状況・動向」といったウイルスゲノム分析・変異株モニタリングができるようになります。
今後の懸念に備えて
今後懸念されていることは、ワクチン接種が広まることによる人流の増大、海外からの新たな変異株の流入やワクチン回避株の出現など。それに伴って、ウイルスゲノム解析をより拡大する必要性があるでしょう。
東大医科研附属ヒトゲノム解析センターとIBMは、今後も引き続き感染拡大防止に貢献できるよう、取り組みを続けるとしています。
(文・和泉ゆかり)
- Original:https://techable.jp/archives/156132
- Source:Techable(テッカブル) -海外・国内のネットベンチャー系ニュースサイト
- Author:izumiyama